技术原理
E3连接酶底物的鉴定可以借助泛素化定量蛋白白组学(quantitative ubiquitination proteomics)来完成。
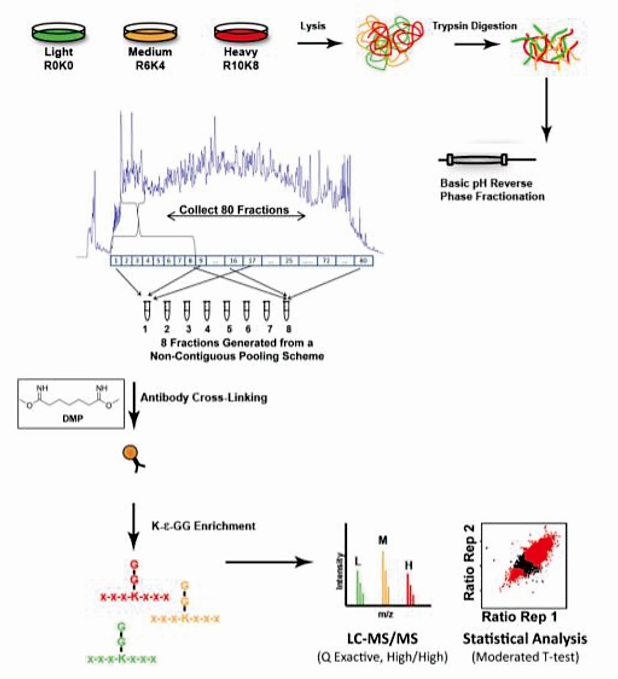
蛋白质底物上泛素化修饰在胰蛋白酶消化后将会将会留下双甘氨酸的标签(K-GG),通过采用特异性识别K-GG的抗体进行富集,并借助高分辨率质谱即可完成对泛素化修饰蛋白的鉴定工作。同时,引入稳定同位素标记培养基中的氨基酸(Lysine),即可定量泛素化修饰的变化,进而筛选E3连接酶底物。
技术优势
1. 可同时比较多个不同处理样品间的泛素化修饰水平变化
2. 一次实验完成对所有泛素化蛋白质及位点的鉴定和相对定量分析
实验流程
参考文献
[1] LA Kane, L Michael, AI Fogel, et al. PINK1 phosphorylates ubiquitin to activate Parkin E3 ubiquitin ligase activity.. J Cell Biol. 2014, 205(2):143-153
[2] G Xu, JS Paige, SR Jaffrey, et al. Global Analysis of Lysine Ubiquitination by Ubiquitin Remnant Immunoaffinity Profiling. Nat Biotechnol. 2010, 28(8):868-873
[3] JV Olsen, B Blagoev, M Mann, et al. Global, In Vivo, and Site-Specific Phosphorylation Dynamics in Signaling Networks. Cell 2006, 127(3): 635-648














