一、研究背景
二、研究手段
1、腔隙性脑卒中PWAS的发现和复制
PWAS鉴定了7个基因(ICA1L、CAND2、ALDH2、MADD、MRVI1、CSPG4和PTPN11)的顺式调节脑蛋白水平与腔隙性卒中相关,4个基因(ICA1L、CAND2、ALDH2和MADD)可以在腔隙性卒中的独立PWAS中复制,提供了较高的置信水平。其他3个不能在确认的PWAS中检测,其中,2个蛋白(CSPG4和PTPN11)没有被测得,MRVI1被测得但没有很大的遗传力,这可能是样本量较小的原因(图1,表1)。
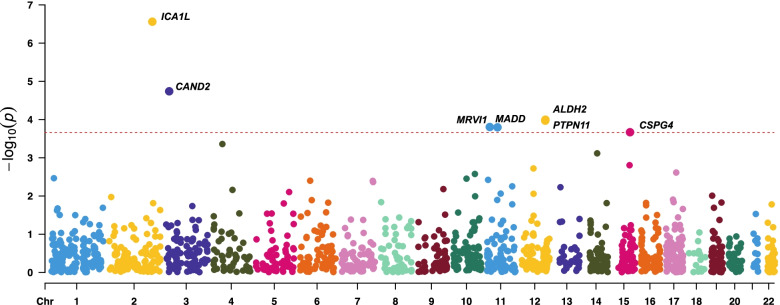

表1
2.脑细胞类型特异性分析
为研究PWAS识别的风险基因是否富集在特定的脑细胞类型中,使用来自Cell Types数据库的人类单细胞RNA-seq数据进行分析。结果表明,7个致病基因的表达存在细胞类型特异性富集,MRVI1和ALDH2在星形胶质细胞中更丰富,而ICA1L、PTPN11和MADD仅在谷氨酸能神经元中发现,GABA能神经元中CAND2和ALDH2水平较高(图2)。

3.MR通过脑pQTL验证4个与腔隙性卒中相关的基因
MR通过脑pQTL进一步证实这些生物标志物(ICA1L、CAND2、ALDH2和MADD)是揭示与腔隙性卒中GWAS相关的有力证据(表2)。

表2
4.脑腔隙性卒中风险基因与pQTL的共定位
基于H4≥75%和PP4/(PP3+PP4)≥0.75,为七个基因中的ICA1L、CAND2和ALDH2提供了基因共定位的证据(表2)。这表明这三个蛋白质在腔隙性卒中的病理生理中发挥重要作用。
5.腔隙性卒中PWAS结果的特异性
为了解腔隙性卒中PWAS结果是否具有特异性,对其他脑相关生物学特征进行PWAS,并预测重要基因的重叠程度与其遗传关系大致相关。缺血性脑卒中PWAS鉴定出4个基因,而大动脉粥样硬化性脑卒中和脑微出血PWAS均未鉴定出。腔隙性脑卒中PWAS发现1/4 (ALDH2;25%)缺血性脑卒中基因与7个腔隙性脑卒中PWAS显著基因重叠,反映了两者高度的遗传相关性。大动脉粥样硬化性中风、脑微出血和腔隙性中风之间没有基因重叠(图3)。

图3 PWAS结果特异性分析
6.基于mRNA水平检测潜在的腔隙性卒中致病蛋白
将腔隙性卒中GWAS数据与人脑转录组相结合,使用FUSION进行转录组全关联研究(TWAS)。结果表明,顺式调控的7个基因的脑mRNA表达与腔隙性卒中相关,值得注意的是,ICA1L在PWAS发现中被确定,这是PWAS和TWAS在腔隙性卒中病因学中发挥作用的联合证据(表3)。
表3
四、结论
总之,研究结果支持三种新的脑蛋白(ICA1L, CAND2和ALDH2)与腔隙性卒中相关,且ICA1L在mRNA水平上得到进一步验证。这些发现为腔隙性脑卒中的遗传和生理过程提供了信息,有利于识别新的治疗靶点。
参考文献:
Zhang C, Qin F, Li X, Du X, Li T. Identification of novel proteins for lacunar stroke by integrating genome-wide association data and human brain proteomes. BMC Med. 2022 Jun 23;20(1):211.














